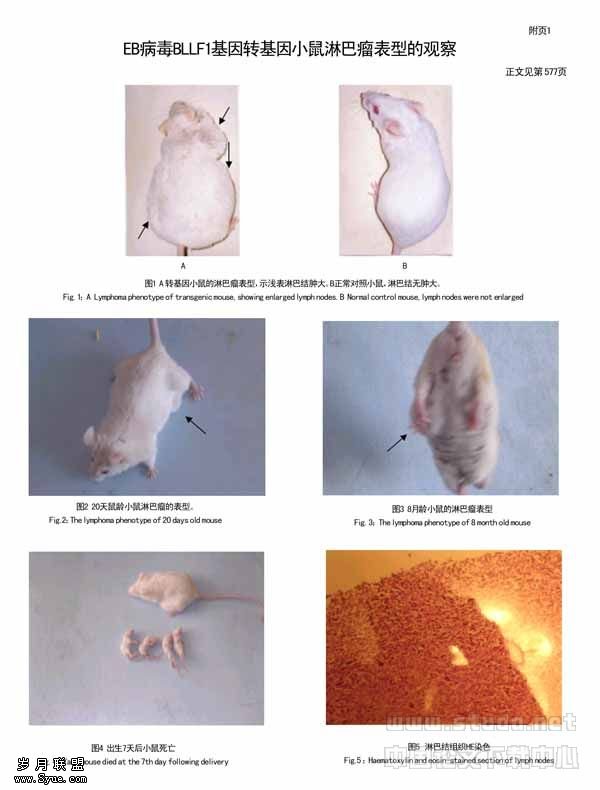
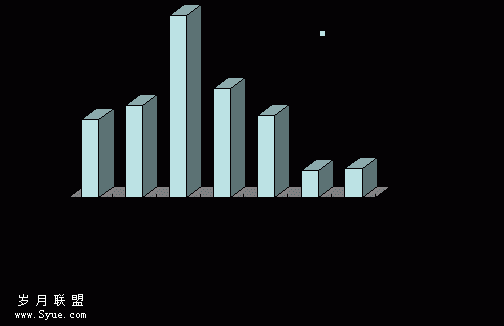丙型肝炎病毒1b型5′端5223 bp半基因组扩增
作者:王小红,熊瑜琳,李俊刚,谭朝霞,胡亚君
【关键词】 丙型肝炎病毒
Amplification of 5′ terminal 5223 bp fragment of hepatitis C virus genotype 1b
【Abstract】 AIM: To amplify 5′ terminal 5223 bp fragment of hepatitis C virus (HCV) genotype 1b. METHODS: Five human serum specimens infected with HCV 1b were obtained from HCV RNA positive repository. Total RNA was extracted by using 3 different methods including TRIzol LS Reagent, QIAamp viral RNA mini kit and Magnetic bead. The extracted RNA was reversely transcribed with 3 reverse transcriptases under different reaction conditions. Optimization for long PCR amplification was performed by comparing different Taq enzymes and cycle systems. The 5′ half of the HCV1b genome was amplified with the refined long RTPCR technique. RESULTS: The integrity of long RNA templates was achieved with TRIzol LS Reagent. Using ReverTra AceaTM and SuperScriptTM ⅡRNase H- reverse transcriptase, the complete cDNA molecules were made with incubation at 42℃ for 10 min followed by 30 cycles of 42℃ for 2 min and 48℃ for 2 min and then 75℃ for 10 min. Long PCR amplification of HCV fragment was established by using TaKaLa LA TaqTM or Platinum Taq DNA polymerase High Fidelity as well as the cycling condition of denaturation for 2 min at 94℃ followed by 30 cycles of 30 s at 94℃, 30 s at 62℃, and 4.5 min at 72℃, or 5 cycles of 15 s at 94℃, and 5 min at 68℃ followed by 20 cycles of 15 s at 94℃, and 5 min with the increment of 10 s per cycle at 68℃ and finally 14.5 min at 72℃. Both cycling conditions were feasible, but amplification efficiency of TaKaLa LA TaqTM is superior to Platinum Taq DNA polymerase High Fidelity under this reaction condition. 5223 bp HCV fragment was obtained from all 5 specimens by utilizing the optimized RTPCR protocol and their genotypes were confirmed by sequence analysis. CONCLUSION: The 5′ terminal 5223 bp fragment of HCV was successfully achieved, which should provide a basis for further construction of fulllength cDNA clone and replicon of HCV 1b.
【Keywords】 HCV; long RTPCR; hemigenome
【摘要】 目的:扩增丙型肝炎病毒(HCV) 1b型5′端5223 bp半基因组. 方法:取5份HCV 1b血清标本,采用3种RNA提取方法,比较几种逆转录酶及逆转录条件,选取不同Taq酶进行长链RTNested PCR反应体系及循环条件的筛选和优化,利用最佳的模板提取方法及优化的RTPCR反应条件扩增出5′端长度为5223 bp(5223 bp)半基因组. 结果:Trizol法可以获得完整的RNA模板,适合长链的扩增;应用ReverTra AceaTM酶和SuperScriptTMⅡ,反应条件为42℃ 10 min后,42℃ 2 min,48℃ 2 min,30个循环,然后75℃,10 min;可获得完整的cDNA模板;TaKaLa LA TaqTM和Platinum Taq DNA polymerase High Fidelity采用两种扩增条件:①94℃变性2 min,94℃ 30 s,62℃ 30 s,72℃ 4.5 min,扩增30个循环;②94℃变性2 min,94℃ 15 s,68℃ 5 min,扩增5个循环,然后94℃ 15 s,68℃ 5 min(每个循环延长10 s), 扩增20个循环,最后72℃延伸10 min,均可获得HCV 1b型5′端5223 bp半基因组的阳性结果,TaKaLa LA TaqTM扩增效果最好;PCR产物经核苷酸序列测定证实为HCV 1b型. 结论:通过各种条件的优化,成功地实现了5′端半基因组的扩增,为下一步全长质粒及复制子的构建打下了基础.
【关键词】 丙型肝炎病毒;长链RTPCR;半基因组
0引言
丙型肝炎病毒(hepatitis C virus, HCV)基因组是一单股正链RNA,全长大约由9600核苷酸组成,是引起丙型肝炎的病原体. HCV基因组具有明显的异质性,根据其核苷酸序列的差异,分为6个主要的基因型,50多个亚型及不同的HCV分离株[1]. 同时,HCV以一群不同的但密切相关的变异株存在于患者体内,称为准种(quasispecies). 传统的HCV全基因组质粒的构建通常采用短片段拼接而成[2],国内有报道采用4个片段扩增构建全长质粒[3],最近国外报道的全长基因组质粒的构建也多是采用3个片段拼接而成[4]. 这种短片段的拼接很容易造成准种间的不同变异株的不真实连接,使所构建的HCV全基因组并不是实际存在的基因,限制了HCV分子水平的研究. 长链扩增可以最大限度地避免这种准种间不真实连接,使所构建的HCV全基因组质粒更为真实可靠. 我们选取HCV主要流行株1b型[5]为研究对象,建立了HCV长链RTPCR扩增方法,并获得了HCV 1b型5′端5223 bp半基因组,为下一步构建病毒全基因组质粒及中国人HCV复制子奠定了基础.
1材料和方法
1.1材料HCV 1b型血清标本5份来自本科HCV标本库,RNA水平为2.966×109~1.251×1010拷贝/L. QIAamp viral RNA mini kit(Qiagen公司);TRIzol LS Reagent,RNaseOUT RNA酶抑制剂, SuperScriptTM Ⅱ反转录酶,Platinum Taq DNA polymerase High Fidelity,RNase H(Invitrogen公司);Viral Purification Kit (MagExtractor,YOYOBO公司); DEPC处理水(上海生工生物技术有限公司);Blue/Orange 6×loading Dye(Promega公司);GeneRuler 1 kb DNA Ladder, readytouse(Fermentas公司);去离子水采用美国Millipore公司的MilliQ plus超纯水仪制造;日立55P72低温超高速离心机(Hitachi公司); PTC225 Peltier Thermal Cycker 热循环仪(MJ RESEARCH公司);Gel Doc2000凝胶成像仪(BioRad公司);试验所需的全部耗材均用美国Axygen公司生产的去DNA酶和去RNA酶处理过的耗材.
1.2方法
1.2.1引物设计根据GenBank 中所有已知HCV1b型全基因组序列,参照文献[6],选择相对保守、GC含量适当的区域,设计一条扩增HCV全长cDNA模板的RT引物和两套巢式PCR正反向引物,扩增产物长度为5258 bp(5223 bp). 同时在C基因的羧基端至E1基因的氨基端(CE1)和3′端区域分别设计了两套用于鉴定RNA模板完整性的巢式PCR正反向引物. 所有引物均由中国上海生工生物工程有限公司合成,PAGE纯化,以DEPC处理水溶解,浓度均为10 μmol/L. 引物的序列和位置见表1.
1.2.2HCV RNA的提取取140 μL血清标本,采用Trizol试剂,QIAamp viral mini kit及Viral Purification Kit(磁珠法),分别按照各自的试剂盒说明书进行病毒RNA的提取.
1.2.3逆转录反应选用ReverTra AceaTM、无RNaseH活性的逆转录酶SuperScriptTMⅡ以及RNA LA PCRTM Kit(AMV) Ver.1.1中的AMV反转录酶进行逆转录反应. 循环条件如表2.
1.2.4cDNA模板完整性的鉴定采用CE1区及3′ UTR的内外引物对长链RT产物的CE1区及3′ UTR进行常规的短链巢式PCR扩增,以鉴定cDNA模板的完整性.
表1HCV RNA 5′端扩增所用引物的序列和位置(略)
*引物位置参照HCV con1株(GenBank注册号为AJ238799).
表2逆转录的反应条件(略)
*: RNA混合液65℃预温5 min立即加入42℃的反应液,然后进行RT反应.
1.2.5长链PCR反应选用Platinum Taq DNA polymerase High Fidelity, TaKaLa LA TaqTM, KOD Dash三种Taq酶进行长链巢式PCR扩增,按说明书配置反应体系. 采用两种循环条件,分别为① 94℃变性2 min后,94℃ 30 s, 62℃ 30 s , 72℃ 4.5 min, 30个循环;②94℃变性2 min后,94℃ 15 s, 68℃ 5 min, 5个循环,继之94℃ 15 s, 68℃ 5 min(每个循环延长10 s), 20个循环,最后72℃延伸10 min. 取4 μL PCR产物进行8 g/L琼脂糖凝胶电泳.
1.2.6PCR产物的纯化和序列测定扩增的PCR产物由上海生工生物技术有限公司完成纯化和序列测定. 序列测定采用ABI公司的3700测序仪自动测序,测序引物为CSI843.
2结果
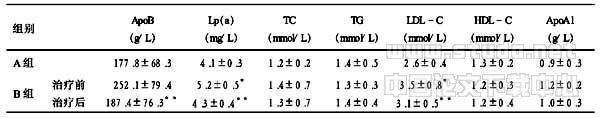
2.1长链RNA模板提取方法的筛选提取的5份血清标本RNA中,Trizol法提取的RNA进行CE1及3′ UTR的扩增均可获得阳性条带. QIAamp viral mini kit提取的RNA有2份标本同时获得阳性条带,其余均为CE1扩增阳性,3′ UTR扩增阴性. 磁珠吸附法提取的RNA均为CE1区扩增阳性,3′ UTR扩增阴性. 说明Trizol提取的模板3′ UTR的poly U结构未破坏,完整性好,适用于长链的扩增(图1).
M:DNA marker ladder (100 bp);1~3,5,6:HCV CE1区扩增产物;7~9,11,12:HCV 3′ UTR扩增产物;4,10:阴性对照;A:TRIzol 法;B:Qiagen RNA抽提试剂盒;C:磁珠法.
图1三种方法提取的RNA模板完整性鉴定(略)
2.2HCV长链RT方法的建立应用ReverTra AceaTM酶和SuperScriptTMⅡ,反应条件为42℃ 10 min后,42℃ 2 min,48℃ 2 min,30个循环,然后75℃ 10 min. 获得的RT产物进行5′端半基因组扩增都获得过阳性结果. 但SuperScriptTMⅡ反转录的模板进行5′端5223 bp片段扩增的成功率高于ReverTra AceaTM酶.
2.3长链PCR方法的建立采用长链nested PCR方法,选用TaKaLa LA TaqTM,在上述两种循环条件下,5份标本均可获得阳性条带(图2),Platinum Taq DNA polymerase High Fidelity只有2份标本获得阳性条带,而ReverTra DashTM未获得阳性条带.
2.45′端5223 bp片段的获得利用上述优化条件,5份HCV血清标本均获得5223 bp长度的目的片段(图2),亮度高,未见非特异性扩增. PCR产物送上海生工生物技术有限公司完成核苷酸序列测定,通过与GenBank中所有1b型序列的比对及系谱分析显示扩增产物为HCV 1b型.
M:DNA marker;1,2,4~6: 血清标本;3:阴性对照.
图2优化的长链RTPCR法对HCV1b型5223 bp半基因组扩增产物电泳图(略)
3讨论
影响长RTPCR成功的因素主要有病毒载量、引物、RNA提取的质量、模板的二级结构、逆转录条件及PCR扩增系统等. 由于HCV不能体外分离培养、患者血清中HCV浓度较低、HCV二级结构复杂及GC含量高等原因使其长链的扩增比较困难. 在实验条件的优化过程中,我们发现以下几点对于成功实现HCV长片段的扩增尤为重要. ①模板RNA的制备:一定数量的完整模板是实现长链扩增的基础. 由于患者血清中RNA含量低,获得高浓度的模板有一定的困难. 国内有报道采用5 mL血清进行HCV RNA的提取用于HCV长链扩增,以保证获得浓度较高的完整模板[3]. 我们仅用140 μL血清,采用Trizol法获得了完整的模板,保障了长链的扩增. ②反转录:常用的逆转录酶如AMV,MMLV等具有较强的RNA酶、H酶活性,逆转录合成长片段第1链cDNA非常困难,限制了RTPCR扩增. 无RNA酶及H酶活性的逆转录酶SuperScriptTMⅡ的应用大大增加了逆转录合成第1链cDNA的长度.
SuperScriptTMⅡ可以在55℃保持酶活性,是公认的逆转录效率较高的酶[6]. HCV 5′和3′ UTR复杂的二级结构不完全变性会阻碍逆转录的进行,升高逆转录的温度有利于打开二级结构[6-7]. ③引物:引物决定PCR扩增产物的特异性和长度. 我们比较了GenBank中所有HCV1b型的全基因组序列,设计了两套5′端半基因组的扩增引物. 设计时避免了形成二级结构及引物二聚体. 在长链扩增中,引物3′端核苷酸的特异性对长链的扩增尤为重要,应与模板严格互补,且与靶序列特异性高[7]. ④酶的选择:选择扩增长链DNA聚合酶的主要标准是具有高保真效果和扩增效率[2,6-7]. 常规PCR应用的耐热DNA聚合酶(如Taq酶等) 具有很强的延伸能力,但由于缺乏3′~ 5′外切酶的活性,不能够校正扩增产物3′端的错配碱基,影响了合成链的近一步延伸. 具有 3′~5′外切酶活性的酶如Pfu,Vent,Deep Vent等,虽然有校读功能,但延伸能力较弱. 长链PCR技术则将两种DNA聚合酶联合应用,利用前者较强的延伸能力和后者的校正功能,通过对缓冲液成分及热循环条件的优化,成功实现了长片段扩增. 在实验中,TaKaLa LA Taq扩增效果最好,通过优化反应条件,5份标本均获得了阳性结果. ⑤循环条件:采用常规的三温循环法即能够实现HCV 5′端半基因组的扩增[6]. 退火温度对PCR反应的特异性影响较大. 退火温度过低会产生非特异性扩增,退火温度过高会降低扩增的效率,因此须合理设定退火温度.
HCV的长链扩增一直是国内外的难点,我们通过各种条件的优化成功的实现了5′端半基因组的扩增,为下一步全长质粒及复制子的构建打下了基础.
【文献】
[1] Simmonds P. Viral heterogeneity of the hepatitis C virus [J]. J Hepatol, 1999,31(1):54-60.
[2] Soo HM, GarzinoDemo A, Hong W, et al. Expression of a fulllength hepatitis C virus cDNA upregulates the expression of CC chemokines MCP1 and RANTES [J]. Virology, 2002,303(2): 253-277.
[3] 毛红霞, 胡芸文, 吴瑛, 等. 丙型肝炎病毒全长cDNA模板的构建及鉴定[J]. 中华实验和临床病毒学杂志, 2004,18(2):122-126.
[4] Sheehy P, Scallanb M, KennyWalsh E, et al. A strategy for obtaining near fulllength HCV cDNA clones (assemblicons) by assembly PCR [J]. J Virol Methods, 2005,123:115-124.
[5] 张帆,王小红,王宇明, 等. 重庆地区HCV基因亚型的分布状态[J]. 第四军医大学报,2005,26(14):1253-1256.
[6] Liu Z, Netski DM, Mao Q, et al. Accurate representation of the hepatitis C virus quasispecies in 5.2kilobase amplicons[J]. J Clin Microbiol, 2004,42(9): 4223-4229.
[7] Lua L, Nakanoc T, Smallwood GA, et al. A refined long RTPCR technique to amplify complete viral RNA genome sequences from clinical samples: Application to a novel hepatitis C virus variant of genotype 6[J]. J Virol Methods, 2005, 126: 139-148.